
 |
||||
 |
||||
|
|
|
Lerneinheit 3: Umsetzung von Gen- in Proteinsequenzen |
|
|
|
|
|
Tätigkeit: In Einheit 2 haben Sie sich mit Verteilungsmustern auf einer Genkarte befasst. Jetzt werden wir Befunde über die DNA-Sequenz nutzen und mit ihrer Hilfe die Sequenz eines Peptids vorhersagen. Jedes Gen ist auf der Genkarte mit einem Pfeil und einer Nummer gekennzeichnet:
Wird es angeklickt, öffnet sich ein Fenster mit der vollständigen DNA-Sequenz des Gens und der daraus abgeleiteten Aminosäuresequenz des Peptids, das es codiert.
1. Probieren Sie es aus. Gehen Sie zur Genkarte für Mycoplasma genitalium und klicken Sie auf das Gen Nummer 042. Daraufhin sollte sich ein neues Fenster mit dem Titel „Dokumentation MG042“öffnen, und Sie sollten folgende Nucleotidsequenz sehen: ttgaacgaacattctttaattgaaattgaaggtttgaacaagacctttgatgatggttat gtttctataagagacattagcctaaatattaaaaaaggcgaatttattactattttaggc ccttctggttgtggtaaaactaccctgttgaggttattagctggatttgaagatcctact tatggcaagatcaaagttaatggtattgacattaaagacatggcaatccataagcgtcct tttgcgacagtttttcaagactatgctttattttcccatctaactgtttataaaaacatt gcttatggtctgaaggtaatgtgaacaaagttagatgaaattccaaaacttgtaagtgat tatcaaaagcaacttgctcttaagcatttaaagctagaaagaaaaatagagcagttacaa aaaaacaattctaatgctcaaagaataaagaaattaaaggaaaaattacaaaaactttta gaaattaacaaacaaaaagttattgagtttgaaaataaagaaaaactacgtagagaagat atttacaagaatttagagcaattaacaaaagaatgggatctactttctcaaaagaaacta aaagaagttgaacaacaaaaacaagcaattgataaaagttttgaaaaagtagagaataaa tacaaaaaagatccttggttttttcaacacagtgaaatacgtttaaaacaatatcagaag aaaaaaactgagttgaaagctgatattaaagcaacaaagaacaaagaacaaatccaaaaa ttaactaaagaacttcaaaccttaaaacaaaaatacgctaataaaaaagcaattgacaaa gagtatgacaaattagttgtagcttacaataagaaagactattgaacttcttattgagaa acatacacacttcaacaaaaagaagcttttgaaaaacgttatctttcaagaaaactaact aaagctgaacaaaataaaaaagttagtgatgttattgaaatggttggtttaaaaggtaaa gaagatcgtttgcctgatgaattatcagggggaatgaaacaaagagttgctttagcacgt tctttagtagtagaacctgaaattcttttattagatgaaccattatctgcacttgatgca aaggttagaaagaatttacaaaaagaattacaacagattcataaaaaaagtggattgact tttatcttagtaactcatgatcaagaagaggctttagttttatcagatcggatagtggtt atgaatgagggaaacatcttacaagttggtaatcctgttgatatttatgactctcctaag actgaatgaattgctaatttcattggtcaagctaacatctttaaaggtacttatttagga gaaaaaaagattcagttacagagtggtgaaatcattcaaactgatgttgataataactat gttgtaggtaagcaatataagatcttaattcgtcctgaagactttgatcttgttcctgaa aataaaggtttttttaatgttcgtgttattgataaaaactacaaaggattgctttgaaag ataaccacacaattaaaagataacactattgttgatttggagagtgttaatgaagttgat gtaaataagacctttggtgttttatttgatcctatagatgttcatttaatggaagtt Diese Sequenz besteht aus sämtlichen Nucleotiden, die ein Peptid codieren, in diesem Fall das „Spermidin-/Putrescintransport-ATP-bindende Protein“. Identifiziert wurde es aufgrund seiner Ähnlichkeit mit einem Gen des gut untersuchten Darmbakteriums Escherichia coli. Das Peptid gehört zu einem Komplex von Membranproteinen, die Spermidin- und Putrescinmoleküle gegen ein Konzentrationsgefälle transportieren und deshalb ATP benötigen. (Der Name „Putrescin“ leitet sich übrigens von putrid ab, dem englischen Wort für „stinkend“.) Das Putrescin dient in der Zelle zum Transport von Aminogruppen, die beim Abbau von Proteinen entstehen, und riecht so unangenehm wie kaum eine andere Verbindung. Es ähnelt stark einem anderen Protein namens "Cadaverin"“ - woher dieser Name kommt und wie das Protein riecht, können Sie sich sicher vorstellen!) Die tatsächliche Aminosäuresequenz können sie anhand des genetischen Codes in der folgenden Tabelle ableiten:
Welches sind die ersten vier Aminosäuren? ____________________ Wenn Sie sich bei der Antwort nicht sicher sind, klicken Sie hier.. Dreibuchstabige Abkürzungen wie Ala-Thr-Ser... erinnern zwar an die Namen der Aminosären, sind aber umständlich, wenn man lange Aminosäuresequenzen aufschreiben will. In wissenschaftlichen Veröffentlichungen werden deshalb häufig Abkürzungen aus nur einem Buchstaben verwendet. Dafür gilt folgende Tabelle:
Wie lauten die einbuchstabigen Abkürzungen für die ersten vier Aminosäuren des Peptids, das vom Gen MG042 codiert wird?_____________
Beachten Sie, dass; die Dokumentation MG042 die vollständige, der DNA-Sequenz entsprechende Peptidsequenz aufführt: LNEHSLIEIEGLNKTFDDGYVSIRDISLNIKKGEFITILGPSGCGKTTLLRLLAGFEDPT YGKIKVNGIDIKDMAIHKRPFATVFQDYALFSHLTVYKNIAYGLKVMWTKLDEIPKLVSD YQKQLALKHLKLERKIEQLQKNNSNAQRIKKLKEKLQKLLEINKQKVIEFENKEKLRRED IYKNLEQLTKEWDLLSQKKLKEVEQQKQAIDKSFEKVENKYKKDPWFFQHSEIRLKQYQK KKTELKADIKATKNKEQIQKLTKELQTLKQKYANKKAIDKEYDKLVVAYNKKDYWTSYWE TYTLQQKEAFEKRYLSRKLTKAEQNKKVSDVIEMVGLKGKEDRLPDELSGGMKQRVALAR SLVVEPEILLLDEPLSALDAKVRKNLQKELQQIHKKSGLTFILVTHDQEEALVLSDRIVV MNEGNILQVGNPVDIYDSPKTEWIANFIGQANIFKGTYLGEKKIQLQSGEIIQTDVDNNY VVGKQYKILIRPEDFDLVPENKGFFNVRVIDKNYKGLLWKITTQLKDNTIVDLESVNEVD VNKTFGVLFDPIDVHLMEV Wie lauten die vollständigen Namen der letzten vier Aminosäuren in dem Protein? __________, ____________, ____________, _____________
2. Beschäftigen Sie sich mit vier beliebigen Genen aus der Genkarte von Mycoplasma genitalium und klicken Sie dazu auf die zugehörigen Pfeile. Benennen Sie mit den einbuchstabigen Abkürzungen die fünf ersten und die fünf letzten Aminosäuren des Peptids, das von dem jeweiligen Gen codiert wird. Nennen Sie außerdem die ersten und die letzten drei Basen.
Gen Nr. ____erste fünf Aminosäuren: ________ erste drei Basen: ____
letzte fünf Aminosäuren: ________ letzte drei Basen: ____
Gen Nr. ____ erste fünf Aminosäuren: ________erste drei Basen: ____
letzte fünf Aminosäuren: ________ letzte drei Basen: ____
Gen Nr. ____erste fünf Aminosäuren: ________ erste drei Basen: ____
letzte fünf Aminosäuren: ________ letzte drei Basen: ____
Gen Nr. ____ erste fünf Aminosäuren: ________ erste drei Basen: ____
letzte fünf Aminosäuren: ________ letzte drei Basen: ____
Gen Nr.____ erste fünf Aminosäuren: ________ erste drei Basen: ____
letzte fünf Aminosäuren: ________ letzte drei Basen: ____
Jetzt prüfen Sie, ob Sie in Ihren Befunden irgendwelche Regelmäßigkeiten finden.
Wenn ja, wie sehen sie aus? ____________
Jetzt überprüfen Sie Ihre Hypothese an den Genen MG001 und MG099 (Klicken Sie dazu auf die Genkarte von Mycoplasma genitalium).
3. Bisher haben wir uns nur die DNA-Sequenzen angesehen, die Informationen für den Aufbau von Peptiden enthalten. Nehmen wir nun einmal an, wir wollten uns das Umfeld eines DNA-Abschnitts außerhalb des Start- und Stopcodons ansehen. Gibt es dazu eine Möglichkeit?
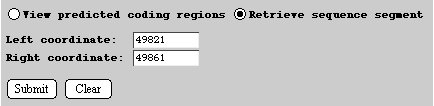
Ja: Hier ist das gesamte Genom gespeichert, und wir können uns jeden beliebigen Teil davon ansehen. Aber denken Sie daran: Die gesamte Sequenz besteht aus 580070 Basen. Damit Sie sich eine Vorstellung davon machen können, was für eine gewaltige Datenmenge das ist, betrachten Sie den folgenden Abschnitt: ttgaacgaacattctttaattgaaattgaaggtttgaacaagacctttgatgatggttat Diese Sequenz besteht aus genau 60 Basen, das heißt, das gesamte Genom wäre 580070/60 = 9668 derartige Zeilen lang! Wenn Sie möchten, können Sie sich weiter mit der Sequenz befassen und dazu die gesamte Sequenz ansehen. Die Datei braucht einige Zeit zum Laden, und deshalb lohnt sich die Mühe vielleicht nicht. In der folgenden Übung werden wir kleine Stücke davon betrachten. Rufen wir einmal einen kleinen Teil der DNA-Rohsequenz aus der TIGR Seite für Einzelpositionen und Abschnitte ab. Die Basen sind von 1 bis 580 070 durchnummeriert. Zunächst suchen wir den Anfang des Gens MG042, von dem zuvor bereits die Rede war. Wir müssen der Datenbank mitteilen, dass wir die DNA-Sequenz zwischen zwei von uns ausgewählten Positionen abrufen möchten, und aus der Dokumentation für MG042 wissen wir, dass das Gen an der Position 49841 beginnt. Geben wir die Positionen 49821 und 49861 ein, so finden wir jeweils 20 Basen beiderseits des Startcodons. Achten Sie darauf, dass Sie die unten aufgeführten Einstellungen genau übernehmen, und drücken Sie dann auf "Abschicken"(Submit). |
|
|
|
|
|
|
||
|
|
|
Jetzt müssten Sie eine DNA-Sequenz von etwa 40 Basen sehen. Erscheint etwas anderes auf Ihrem Bildschirm, haben Sie den Knopf "Abrufen von Sequenzabschnitten" nicht richtig eingestellt. Überprüfen Sie an dem obigen Schema, ob Ihre Einstellungen damit übereinstimmen, und probieren Sie es dann noch einmal.
Schreiben Sie hier Ihre abgerufene DNA-Sequenz auf: _____________________________________________________
Vergleichen Sie jetzt diesen Abschnitt mit der Sequenz am Anfang des Gens MG042, den Sie oben in Schritt 1 herausgesucht hatten: 5'-ttgaacgaacattctttaatt-3'......Sie sollten dann in der Lage sein, die sogenannte Startsequenz zu finden, die zum Genanfang passt. Die letzte Base des Gens MG042 befindet sich in der Position 51517. Suchen Sie mit Hilfe der Seite für Einzelpositionen und Sequenzabschnitte die DNA-Sequenz auf, die sich beiderseits dieser Endposition zehn oder zwanzig Basen weit erstreckt.
Schreiben Sie diese Nucleotidsequenz hier auf:
Jetzt vergleichen Sie sie mit dem codierenden DNA-Abschnitt des Gens MG042: .....5'-atttgatcctatagatgttcatttaatggaagtt-3'
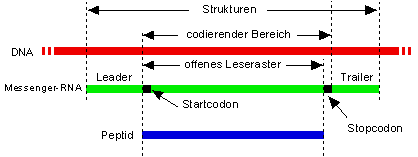
4. Wir müssen jetzt kurz innehalten und uns etwas genauer mit dem Begriff des Gens befassen. In dieser Datenbank werden Peptide und die zugehörigen DNA-Sequenzen, die sie codieren, mit Nummern (001,002 usw.) bezeichnet. In unserem Beispiel ist MG042 als die DNA-Sequenz definiert, die ein bestimmtes Peptid codiert. Vermutlich ist Ihnen aber bereits aufgefallen, dass in der Dokumentation für MG042 kein Stopcodon aufgeführt ist; es besteht aus den nächsten drei Basen. Können wir demnach MG042 als Gen bezeichnen, oder ist es etwas anderes? In der Legende zur Dokumentation für MG042 heißt es: "Ende 5 und Ende 3 sind die Koordinaten des offenen Leserastern im Genom von M. genitalium." Was ist ein offenes Leseraster, und was hat es mit dem Gen zu tun? Ein offenes Leseraster (open reading frame, ORF) ist definiert als ein Startcodon (ATG), gefolgt an irgendeiner Stelle von einem Stopcodon in dem gleichen Leseraster. Die folgende Sequenz ist demnach ein ORF: atgxxxyyyzzztag... (wobei xxxyyyzzz beliebige Basen repräsentiert) Die Ribosomen lesen, beginnend beim Startcodon, eine Basen-Dreiergruppe nach der anderen ab: atg, xxx, yyy, zzz, tag = stop. Die folgende Sequenz dagegen ist kein offenes Leseraster, obwohl sie ebenfalls die Buchstaben atg und tag enthält: atgxxxyyyzzzztag... Liest man hier Dreiergruppen ab, trifft man nicht auf ein Stopcodon: xxx, yyy, zzz, zat, g...
Welche der folgenden Sequenzen enthalten ein ORF? (Ein Tip: Suchen Sie vom 5’-Ende aus nach dem ersten Startcodon. Zählen Sie dann jeweils drei Basen ab und achten Sie auf Stopcodons.)
Die Definition eines Gens ist komplizierter. Wie Sie bereits wissen, muss die DNA zunächst in RNA transkribiert werden, bevor ihre Information in ein Peptid umgesetzt werden kann. Das RNA-Transkript enthält beiderseits des offenen Leserasters noch Basensequenzen, die nicht in Peptidsequenzen umgeschrieben werden und die man deshalb als Leader- und Trailersequenzen bezeichnet. Ein Strukturgen kann man definieren als die gesamte DNA-Sequenz, die in RNA transkribiert wird; der Teil der Sequenz, der tatsächlich in ein Peptid translatiert wird, ist das offene Leseraster. |
|
|
|
|
|
|
||
|
|
|
Betrachten Sie noch einmal die Genkarte für Mycoplasma genitalium. Entsprechen die Pfeile, die mit MG001, MG002 usw. bezeichnet sind, den Genen oder den ORFs? ________
5. Eines der kleinsten ORFs von Mycoplasma genitalium ist MG174. Es codiert das kleinste Protein in den Ribosomen der Zelle. Seine vollständige DNA-Sequenz besteht nur aus den folgenden 111 Buchstaben: atgaaggttagagcaagcgtaaaaccaatttgtaaagattgtaagatcatcaaacgtcac cgcatcttaagggtgatctgcaaaaccaaaaaacacaagcaaaggcaagga Wieviele Aminosäuren codieren diese DNA?______ Schreiben Sie die vollständige Aminosäuresequenz des Proteins mit den einbuchstabigen Abkürzungen auf. _______________________________________________ Um zu überprüfen, ob Sie es richtig gemacht haben, klicken Sie in der Genkarte von Mycoplasma genitalium auf den Pfeil für MG174.
|
|
|