
 |
||||
 |
||||
|
|
|
Untersuchung des Bakteriengenoms Lernaktivität 3: Umsetzung von Gen- in Proteinsequenzen Name ___________________________ Kurs ___________________________ Datum ___________________________
Beantworten Sie die folgenden Fragen:
Welches sind die ersten vier Aminosäuren? ____________________
Wie lauten die einbuchstabigen Abkürzungen für die ersten vier Aminosäuren des Peptids, das vom Gen MG042 codiert wird? _____________ Wie lauten die vollständigen Namen der letzten vier Aminosäuren in dem Protein? __________, ____________, ____________, _____________
2. Suchen Sie sich fünf Gene aus, die Sie genauer untersuchen möchten. Klicken Sie in der Genkarte für for Mycoplasma genitalium auf die entsprechenden Pfeile. Nennen Sie die ersten und letzten fünf Aminosäuren der Peptide mit ihren einbuchstabigen Abkürzungen. Nennen Sie außerdem jeweils die ersten und letzten drei Basen. |
|
|
|
|
Gen Nr. ____ erste 5 Aminosäure: ________erste drei Basen: ____
letzte fünf Aminosäuren:________ letzte drei Basen: ____
Gen Nr. ____erste 5 Aminosäuren: ________ erste drei Basen: ____
letzte fünf Aminosäuren: ________ letzte drei Basen: ____
Gen Nr. ____ erste 5 Aminosäuren: ________erste drei Basen: ____
letzte fünf Aminosäuren: ________ letzte drei Basen: ____
Gen Nr. ____ erste 5 Aminosäuren: ________erste drei Basen: ____
letzte fünf Aminosäuren: ________ letzte drei Basen: ____
Gen Nr. ____ erste 5 Aminosäuren: ________erste drei Basen: ____
letzte fünf Aminosäuren: ________ letzte drei Basen: ____
|
|||
|
|
|
Jetzt prüfen Sie, ob Sie in Ihren Befunden irgendwelche Regelmäßigkeiten finden.
Wenn ja, wie sehen sie aus?____________
Wenn ja, wie sehen sie aus?____________
Erläutern Sie, warum: _______________________________________________ |
|
|
|
|
|
|||
|
|
|
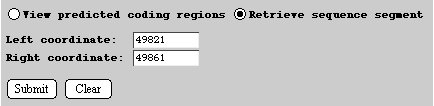
Jetzt müßten Sie eine DNA-Sequenz von etwa 40 Basen sehen. Erscheint etwas anderes auf Ihrem Bildschirm, haben Sie den Knopf "Abrufen von Sequenz Abschnitten" nicht richtig eingestellt. Überprüfen Sie an dem obigen Schema, ob ihre Einstellungen damit übereinstimmen, und probieren Sie es dann noch einmal.
Schreiben Sie hier ihre abgerufene DNA-Sequenz auf: _____________________________________________________
Vergleichen Sie jetzt diesen Abschnitt mit der Sequenz am Anfang des Gens MG042, den Sie oben im Schritt 1 herausgesucht hatten: 5'-ttgaacgaacattctttaatt-3'......Sie sollten die Sequenz finden können, die dem Anfang des Gens entspricht. Die letzte Base des Gens MG042 befindet sich in der Position 51517. Suchen Sie mit Hilfe der Seite für Einzelpositionen und Sequenzabschnitte die DNA-Sequenz auf, die sich beiderseits dieser Endposition zehn oder zwanzig Basen weit erstreckt. Schreiben Sie diese Nucleotidsequenz hier auf: ____________________________________
Jetzt vergleichen Sie sie mit dem codierenden DNA-Abschnitt des Gens MG042: .....5'-atttgatcctatagatgttcatttaatggaagtt-3'
5. Eines der kleinsten ORFs von Mycoplasma genitalium ist MG174. Es codiert das kleinste Protein in den Ribosomen der Zelle. Seine vollständige DNA-Sequenz besteht nur aus den folgenden 111 Buchstaben: |
|
|
|
|
|
atgaaggttagagcaagcgtaaaaccaatttgtaaagattgtaagatcatcaaacgtcac cgcatcttaagggtgatctgcaaaaccaaaaaacacaagcaaaggcaagga | ||
|
|
|
_______________________________________________ |
|
|